尋夢新聞LINE@每日推播熱門推薦文章,趣聞不漏接
180+篇植物基因組文章解讀大全
第四章 豆科
地表最強植物基因組文獻解讀,正在繼續。科技君和小夥伴們特地對植物基因組領域已發的180多篇高質量文章進行收集、解讀和歸類,經歸納整理後共分十章,前九章為相關領域已發表物種文獻解讀,最後一章為植物基因組未來發展趨勢及預測。

導 讀
花生在如今是一種隨處可見的堅果,但在乾隆末年花生還是專屬貴族的食物,尋常百姓家很難吃到。直到100年後,受到西方國家的影響,使用紅薯和花生輪作,這才讓花生和紅薯走進千家萬戶。
該研究完成了栽培花生的二倍體野生種A.duranensis和A.ipaensis的基因組測序,這對四倍體栽培種下一步的測序、分析、拼接具有重要參考價值。同時,野生種具有豐富的抗病、抗逆等優異基因資源,其測序的完成有助於深刻闡明花生的起源與進化,系統解析重要性狀形成的分子基礎,高效發掘控制重要農藝性狀的功能基因,從而推動花生分子設計與聚合育種的開展對於增加花生產量、提高生產效益、保障食品安全、促進產業可持續發展等具有重要的理論價值和應用前景。
文獻題目:The genome sequences of Arachis duranensis and Arachis ipaensis, the diploid ancestors of cultivated peanut
發表期刊:Nature Genetics
發表時間:2016年
影響因子:27.959
摘要介紹:
栽培花生(Arachis hypogaea),異源四倍體,由2個緊密相關的二倍體基因組經過雜交加倍形成,總基因組大小為2.7Gb。因栽培花生基因組龐大而複雜,使得組裝到染色體水平非常具有挑戰性。該研究報導了栽培花生的兩個二倍體祖先種(Arachis duranensis 和 Arachis ipaensis)的基因組序列,為四倍體栽培花生基因組研究提供了基礎。
研究結果表明,這兩個二倍體祖先種基因組分別與栽培花生的A、B亞基因組相似,並能夠用來鑒定候選的抗病基因、引導四倍體轉錄本的拼接和檢測栽培花生亞基因組之間的基因交換。另外,基於A.ipaensis和栽培花生B亞基因組DNA的高度相似性和生物地理學證據表明,研究者推斷A.ipaensis可能是與栽培花生B亞基因組供體相同群體的直接後代。
內容簡析
研究問題:
1. 栽培花生四倍體的二倍體祖先種基因組結構特徵和甲基化水平差異;
2. 祖先種通過雜交加倍形成栽培花生過程中,基因組層面經歷了哪些變化;
3. 二倍體祖先種的進化以及與栽培種之間的分化時間推斷;
4. 栽培花生四倍體轉錄本的組裝;
5. 抗害蟲、抗病基因的篩選和QTL定位。
研究方向:
1. 兩個野生種花生de novo基因組學研究
2. 兩個野生種花生DNA甲基化分析
3. 野生種、栽培種花生轉錄組分析
4. 野生種、栽培種花生比較基因組學分析
研究難點:
栽培花生為異源四倍體,基因組大且重復含量高,這使得直接組裝栽培花生基因組非常具有挑戰性。
研究亮點:
1. 該研究獲得了栽培花生二倍體祖先種基因組序列,並在此基礎上重構了獲得的四倍體栽培花生轉錄本序列;
2. 該研究定位了野生花生種中抗害蟲、抗病相關基因;
3. 該研究通過大量的比較分析,分析A. duranensis和A. ipaensis雜交加倍後形成四倍體栽培花生基因組進化的過程。找到了花生四倍體亞基因組之間存在基因交流的證據,並且分析發現重組都發生在花生四倍體A亞基因組;而花生四倍體B亞基因組與二倍體A. ipanensis DNA水平相似性很高,兩者在大約9,300年前分開。
研究方法
研究對象:
1. 栽培花生的二倍體祖先 (Arachis duranensis 和 Arachis ipaensis);
2. 四倍體栽培花生 (Arachis hypogaea);
所用軟件:
二倍體基因組組裝:COPE、SOAP denovo v2.05、KGF、Gapcloser、SSPACE;
花生四倍體轉錄本分析:FastQC、Trim Galore! v0.3.5、Trinity、Bowtie、RSEM、GMAP、GSNAP、BLAST;
遺傳圖譜構建和組裝結果檢查:Mapmaker Macintosh 2.0、CLC Genomics Workbench、SAMtools、MadMapper、Carthagene;
假染色體連接:BLAST、ePCR (electronic PCR);
重復序列分析:RepeatMasker、LTR_FINDER、BLAST、CAP3、Biolayout、Gepard、Artemis、HMMER、SINE-Finder、MITE-Hunter;
基因預測和功能註釋:MAKER-P流程(SNAP)、InterProScan、BLAST、AHRD;
基因復制分析:Muscle、HMMER;
抗病相關基因和NB-LRR編碼基因分析:HMM、BLASTP、Excel;
基因組進化分析:DAGchainer、PAML (codeml)
染色體結構和共線性分析:CViT、MUMmer(mummer和mummerplot)、DAGChainer;
序列比較分析:MUMmer(nucmer,show-coords,show-tiling)、Excel;
花生RILs遺傳交換分析:FASTX-Toolkit、Bowtie 2、BEDtools、SAMtools、R、Excel;
所用數據:
1. 兩個二倍體祖先種的基因組測序數據(250Bp、500Bp、2Kb、5Kb、10Kb、20Kb以及40Kb);
2. 兩個二倍體祖先種及栽培花生四倍體低深度Moleculo長讀段數據;
3. 兩個二倍體祖先種和栽培花生四倍體各自近親雜交產生後代及親本的GBS測序數據;
4. 兩個二倍體祖先種的甲基化測序數據;
5. 兩個二倍體祖先種和四倍體栽培花生轉錄組測序數據;
所用數據庫:
1. Transposable element protein databases;
2. EST and NCBI;
3. SwissProt and TrEMBL databases;
4. SoyTEdb and Repbase library;
5. Protein databases(Including ProDom、PRINTS、 Pfam、 SMART、 PANTHER and PROSITE);
6. Food and Agriculture Organization Corporate Statistical Database (FAOSTAT);
7. Phytozome 10;
8. Natural Earth maps;
實驗過程:
De novo樣品信息:二倍體花生A. duranensis V14167和 A. ipaensis K30076用於基因組測序;A. duranensis K7988、A. ipaensis K30076和A. hypogaea cv. Tifrunner用於Moleculo建庫測序;
遺傳圖譜:二倍體A基因組群體:A. duranensis K7988和A. stenosperma V10309雜交的F5代90個個體;二倍體B基因組群體: A. ipaensisKG30076和A. magnaKG30097雜交的F6代94個個體。四倍體AB RIL群體是A. hypogaea cv. Runner IAC 886和秋水仙素誘導的A. ipanesis K30076與A. duranensis V14167四倍體雜交的F6代的89個個體。
轉錄組樣品準備:二倍體花生(A. ipaensis和A. duranensis)葉(包括葉柄),莖、根、花、雌蕊柄等5個部位的樣品提取RNA,反轉錄為cDNA。其中根組織提取於3月齡以上的植株,莖組織提取於3月齡以內的植株。
研究結果
研究成果:
1. 基因組測序和組裝
(1) 對插入片段大小為250Bp、500Bp、2Kb、5Kb、10Kb和20Kb的文庫以及fosmid方法構建的插入片段大小為40Kb文庫進行雙末端測序,read長度90-150Bp。共獲得了A.duranensis的325.73 Gb的原始數據和A.ipaensis的416.59 Gb的原始數據,過濾低質量讀段後,測序深度分別為154×和163×。
(2) 基於SOAPdenovo組裝得到A.duranensis基因組大小為1,211Mb,A.ipaensis基因組大小為1,512Mb。基於遺傳圖譜數據,分別將A.duranensis 1,025Mb和A.ipaensis 1,338Mb序列各連接成10條假染色體。
2. 轉座子分析
A.duranensis和A.ipaensis中轉座元件(TE)分別占基因組的61.7%和68.5%,與已報導的栽培花生重復序列比例接近(64%)。兩個物種基因組中大部分TE家族相同,但因轉座子本身特性,他們的位置以及相對豐度不同。Ty3-gypsy和non-LTR中少數的家族豐度非常高,主要集中在中心粒區,這些家族包括FIDEL、Feral、Pipoka、Pipa,以及 新髮現的Apolo和Polo。總體來講,每個基因組中LTR逆轉錄轉座子占一半以上,而DNA轉座子僅占10%。其中,兩個基因組中7.8%和11.7%分別為長散在重復序列(LINE),是迄今為止植物基因組比例最高。
3. 基因註釋和基因重復分析
基於MAKER2流程分別註釋得到A.duranensis和A.ipaensis的36,734和41,840個高質量的編碼基因。基於基因復制分析,結果提示A.ipaensis中基因數量比A. duranensis多主要因其發生局部復制事件,這些基因包括NB-ARC、leucine-rich repeat (LRR)、pentatricopeptide-repeat、kinase、WD40-repeat和kinesin蛋白。
4. DNA甲基化
基於MethylC-SEQ對A.duranensis和A.ipaensis進行DNA甲基化測序,分別得到189,653,337(~8.6×)和277,101,705(~10×)條唯一比對的reads。兩個物種基因組甲基化水平相似,其中CHG位點(H是A,T或C)甲基化分別為57%和60%,CG位點甲基化分別為73%和75%,CHH位點甲基化分別為8%和6%。
5. 抗病相關的NB-LRR編碼基因定位
Nucleotide-binding-leucine-rich repeat (NB-LRR)編碼基因在抵禦害蟲和抗病中有重要作用。在A.duranensis和A.ipaensis中分別鑒定出345和397個NB-LRR編碼基因。該家族基因大部分成簇排列在基因組上,如染色體2遠端,染色體4短臂以及染色體9長臂上。基於QTL分析,抗性相關的基因定位於A. duranensis的染色體3的短臂末端,這其中就包括Aradu.Z87JB基因。
6. A.duranensis和A.ipaensis的物種進化和分化時間推斷
分析表明,自5800萬年前Dalbergioid 分支的物種分化後,花生積累的突變相對較快。A.duranensis和A.ipaensis的旁系同源基因KS值分別為0.95和0.90,相比較荷花(~0.65)、大豆(~0.65)和菜豆(~0.8),該值與苜蓿(~0.95)中KS更接近。根據A. duranensis和A. ipaensis直系同源基因的KS峰值在0.035以及花生的平均變化率為8.12×10-9 KS/年,推測這兩個物種在大約216萬年前分化。
7. 染色體結構和共線性分析
與細胞遺傳學觀察結果一致,大部分假染色體對稱分布在近著絲粒兩側,且這兩個物種中大部分的染色體為1:1的對應關係:兩個物種染色體2、3、4和10為共線性關係,染色體5、6和9主要在一端發生大片段的倒置,染色體1則在兩臂都有大的倒置。而染色體7和8發生複雜的重排,將富含重復序列的片段轉至A基因組的7號染色體,而富含基因區片段則轉至A基因組9號染色體上,使得A基因組的7號染色體僅有一端正常的染色體臂,而8號染色體異常的短,該結果與細胞學觀察結果一致。A. ipaensis假染色體都比A. duranensis相應的染色體長度更長,一部分原因是因局部復制和轉座子多。
點圖結果顯示,共線性的染色體中,A. duranensis80%~90%的區域都相應的在A. ipaensis染色體上。而染色體臂有一端倒置的情況下,點圖中出現不同的弧度,這些弧度是因染色體遠端到中心粒附近的DNA丟失和獲得的速率不同造成的。在染色體沒有倒置區域、基因、重復序列和甲基化水平呈現梯度分布,且從中心粒區域到染色體末端呈下降趨勢。然而,在A. duranensis中,經歷過大面積的重組區域,這些規律則被打斷。因此研究者推斷主要重組都發生在A基因組中。相比較共線性的同源染色體,因大面積重排導致同源染色體長度差異更大。A. duranensis染色體倒置現象較預期小,該結果也支持,相比較DNA積累,DNA清除占主要。
8. 野生種花生和四倍體栽培花生的序列比較分析
(1) 二倍體假染色體與栽培花生連鎖群基本上為一一對應的關係。在三張遺傳圖譜的標記分子中,分別有83%、83%和94%能匹配到假染色體上。將四倍體的2× Moleculo讀段比對到二倍體假染色體上,結果顯示,栽培花生與A. duranensis和A. ipaensis矯正後平均相似性分別為98.36%和99.96%。從下圖6中可以看出,栽培花生A. hypogaea與二倍體B基因組較二倍體A基因組更為相似。
研究者進一步發現A. hypogaeaA和B亞基因組有基因重組信號,與預期相符,這些信號在共線性的同源染色體中更為頻繁。這種重組導致栽培種亞基因組與其相應二倍體基因組序列相似性降低。研究者發現栽培種Moleculo reads比對到A基因組共線性的假染色體序列相似性比比對有倒置的假染色體的相似性更低。這種趨勢在B基因組整個染色體上較弱,但是還是能明顯看出在共線性的B亞基因組染色體臂末端相似性大幅度的降低。
(2) 研究者推斷A. duranensis和A. ipaensis大概在216萬年前分開。A. duranensis V14167與栽培種A基因組大致24.7萬年前分開,而A. ipaensis與栽培種B基因組大致9,400年前分開。
(3) 研究者進一步利用栽培種RILs群體估算A、B亞基因組的重組率,結果顯示,大部分譜系A和B基因組相對劑量是相等的,與親本相同,但RIL028這個譜系相對劑量在同源染色體區域急劇的改變(下圖7,A基因組104-102Mb區域和B基因組112-126Mb區域)。比對到B基因組相對劑量幾乎沒有了,而比對到A基因組的相對劑量則急劇增加。這清楚的表明,栽培花生A和B基因組發生了基因交換,這在四體重組中可能出現,但也可能形成Holiday junction之後發生基因轉換。
9. 二倍體基因組引導的四倍體轉錄組裝
研究者通過三種策略和四種不同的軟件對栽培花生轉錄本拼接並比較每種策略拼接得到的轉錄本準確性。這三種策略分別為:從頭組裝、分出屬於A和B的數據集進行獨立組裝、將A、B假染色體進行整合後引導轉錄本拼接。將不同策略不同軟件得到的轉錄本比對(無錯配)回假染色體上百分比來評價組裝結果的準確性。
結果顯示從頭組裝策略得到的轉錄本準確性最低(32.17~39.82%),其次是分開組裝(40.07~55.8%),最後是在基因組引導組裝結果準確性最高(65.87%)。研究者進一步過濾掉轉座元件、低表達的轉錄本以及去冗餘,最終得到183,062個轉錄本,其中88,643(48.32%)屬於A基因組,94,419(51.58%)屬於B基因組。
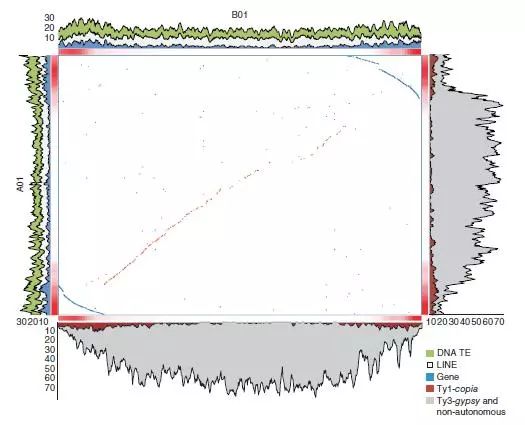
圖1 染色體假分子A01和B01的結構概述和比較
堆疊區域代表基因和轉座元件在染色體上的分布。高重組率(熱圖中紅色)區域集中在染色體末端。在點圖中,發生倒置的區域形成了弧線,表明,自這兩個物種分化之後的進化歷程中,累積的DNA突變在染色體中心區域更多,而DNA清除則在染色體末端區域更加頻繁。基因、DNA轉座元件和Ty1-copia元件主要集中在染色體末端區域。Ty3-gypsy元件主要集中在染色體中心區域。
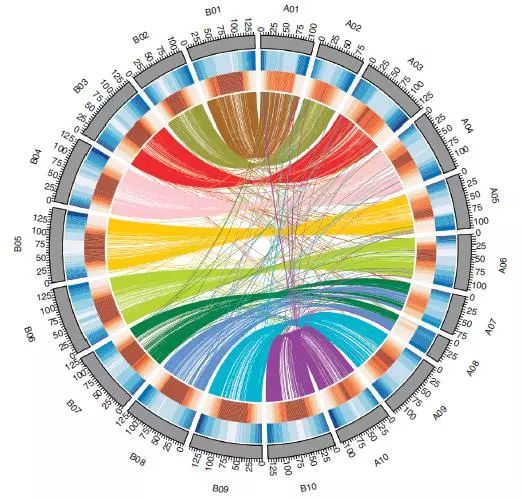
圖2 A. duranensis和A. ipaensis染色體假分子
之間的關係
藍色:基因密度;棕色:Ty3-gypsy元件和非自主LTR反轉錄轉座子的密度;灰色標尺表示以兆為單位。
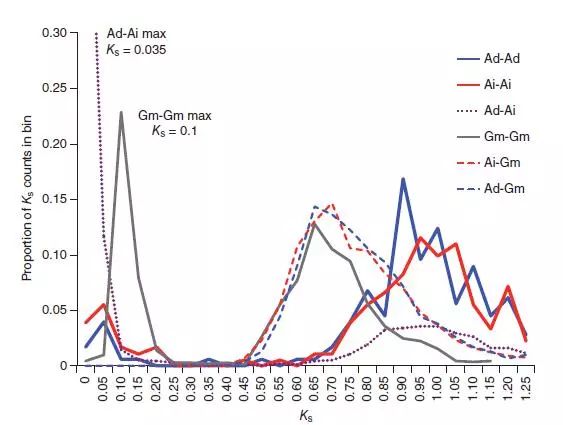
圖3 突變和基因組重復
該圖顯示A. duranensis(Ad)、A. ipaensis(Ai) 和 Glycine max(Gm)的旁系同源基因和直系同源基因的同義突變(Ks)的分布。G.max-G.max比較中的峰值代表大豆在Ks=0.10(~10百萬年)和Ks=0.65(58百萬年)發生全基因組復制事件(WGD)。同樣早期的蝶形科WGD同樣影響了落花生屬,因此A. duranensis–A. duranensis和A. ipaensis–A. ipaensis峰值(分別在Ks=0.90和0.95)的偏移表明花生屬以快於大豆1.4倍的速度累積同義突變。基於花生屬平均突變率為8.12×10-9Ks/年,研究者可能A. duranensis和 A. ipaensis在大約216萬年前分開。
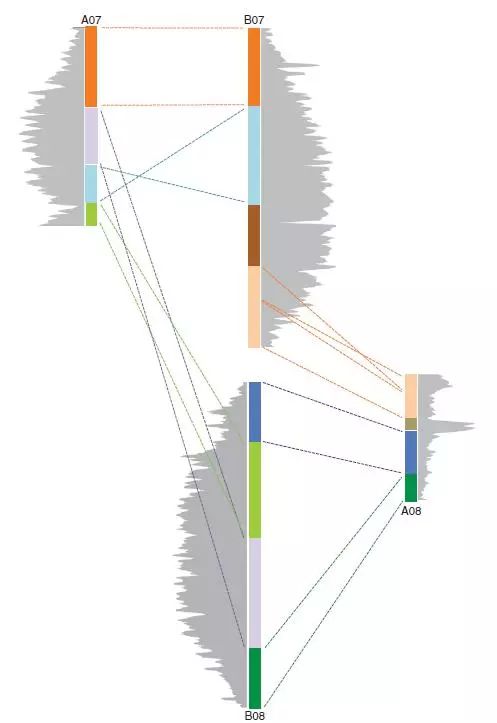
圖4 染色體7和染色體8之間的重排示意圖
重排產生了小片段且重復序列較少的染色體,如A. duranensis8號假染色體、A. duranensis7號假染色體上只有一部分正常的常染色質臂。共線性的染色體片段由相同的色塊表示;Ty3-gypsy元件和非自主反轉座元件的分布用灰色表示;從圖中可以看出,A基因組8號染色體的重復序列比例低且重復序列高的區域(鼓起處)在染色體上部遠端區。
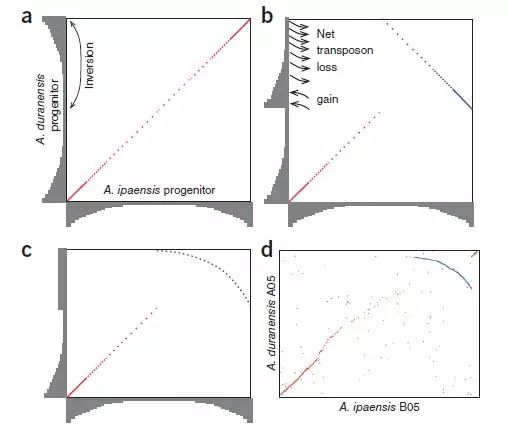
圖5 A和B基因組分歧之後的基因組區域反轉示意圖
灰色:基因密度
a: 倒置將重復序列高、基因密度的DNA轉運到染色體遠端區域,並將重復序列低且基因密度高的DNA轉運至染色體中心的區域;
b: 在染色體遠端區域,倒置的片段通過重組使DNA丟失,而染色體中心區域則獲得DNA;
c: 在a和b的基礎上形成了特徵性弧(藍色點)、不規則的基因、重復序序列和甲基化密度分布特徵。發生這種不規則的模式表明所有主要基因組重組發生在A基因組上。
d: A05和B05的比較分析的點圖顯示特徵弧。
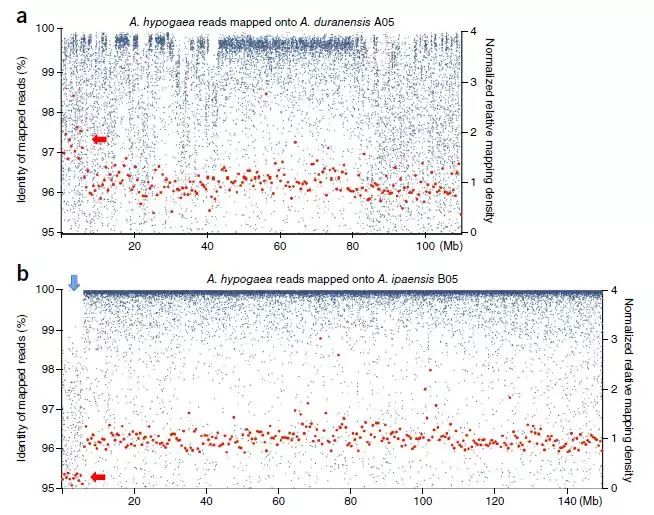
圖6 栽培花生與Arachis duranensis和Arachis ipaensis的假染色體比較結果示例
(a、b)中圖顯示的是栽培種Moleculo 讀段比對到A05和B05的情況。藍色點代表read比對到染色體的相似性百分比,紅色代表0.5Mb滑窗內Moleculo鹼基深度分布。結果顯示,相比較比對上A基因組染色體5遠端區域,比對中心區域的相似性分布更為一致。這可能反應栽培種A亞基因組與A. duranensis因重組導致序列相似性降低。而在B基因組上相似性更高且一致性好,除了染色體上端6.1 Mb的區域,該差異可能提示在這部分區域,栽培種的染色體倍型由AABB變為AAAA。
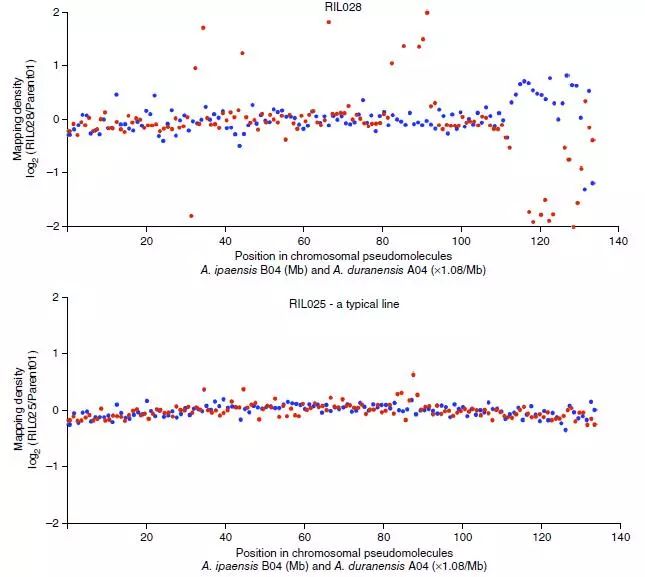
圖7 栽培花生中亞基因組之間遺傳交換的鑒定
上圖提示RIL028中A04和B04之間發生重組的結果;下圖提示RIL025中A04和B04之間沒有發生重組的結果。Y軸表示比對密度,X軸表示比對的位置信息;滑窗為1Mb;從圖中可以看出,在RIL028中,亞基因組的相對劑量在染色體下臂發生較大改變,該結果表明,A和B亞基因組間發生基因交換。
圖8 A. duranensis、A. magna、A. ipaensis以及栽培花生A. hypogaea的起源地分布
【參考文獻】
Bertioli DJ, et al: The genome sequences of Arachis duranensis and Arachis ipaensis, the diploid ancestors of cultivated peanut. Nat Genet 2016, 48(4):438-446.
【數據鏈接】(復制網址後使用瀏覽器打開)
轉座元件蛋白質數據庫:http://www.repeatmasker.org/RepeatProteinMask.html#database
Food and Agriculture Organization Corporate Statistical Database (FAOSTAT):http://faostat3.fao.org/home/
Arachis基因組相關測序數據及分析結果:http://peanutbase.org/download
MethylC-seq數據:Gene Expression Omnibus(GEO),登錄號:GSE71357

「閱讀原文」
,查看文獻!
撰稿:大項目部-尹立新、許蓮
編輯:市場部
▼